Ze stonden lange tijd bekend als een lastige groep; de membraaneiwitten. Vooral hun structuur was moeilijk te achterhalen. Maar de laatste jaren krijgen structuurbiologen steeds meer grip op membraaneiwitten. ’Wat in het verleden als een uitdagend project te boek stond, is nu een gewoon standaardproject.’
Eigenlijk geldt voor membraaneiwitten wat voor elk eiwit geldt: ‘Als je wilt begrijpen hoe het werkt, heb je niet alleen functionele data, maar ook de structuur nodig’, zegt Bert Poolman, hoogleraar biochemie aan de Rijksuniversiteit Groningen. Bij membraaneiwitten komt daar nog bij dat ze de poortwachters van de cel zijn en daarmee vaak het doelwit van medicijnen. Voor de ontwikkeling daarvan helpt het om te kunnen zien hoe die moleculen met het membraaneiwit interacteren.
En ondanks dat machinelearning goede voorspellingen van eiwitstructuren doet, blijft het belangrijk om membraaneiwitten te karakteriseren. ‘Er ontbreekt nog heel veel kennis’, zegt Anastassia Vorobieva, groepsleider computational protein design bij het VIB-VUB Center for Structural Biology. Zo heeft AlphaFold, het AI-systeem dat eiwitstructuren voorspelt, vaak nog moeite met membraaneiwitten. De reden daarachter is dat slechts 2% van de eiwitten in de Protein Data Bank, de database waarmee is AlphaFold getraind, uit membraaneiwitten bestaat.
Kristalvrij
Dat structuurbiologen toch meer grip op membraaneiwitten krijgen is te danken aan twee ontwikkelingen. De eerste is de opkomst van cryo-elektronmicroscopie (Cryo-EM) in de laatste vijftien jaar. Voor die tijd waren structuurbiologen afhankelijk van X-ray kristallografie en het maken van eiwitkristallen. Maar kristallen van in zeepmicellen geplaatste membraaneiwitten zijn moeilijk te maken, fragiel en vallen vaak al uit elkaar voordat onderzoekers aan hun meting beginnen. Met de komst van Cryo-EM zijn kristallen niet meer nodig. ‘Onderzoekers kunnen membraaneiwitten weer terugzetten in een stukje lipide dubbellaag, bijvoorbeeld een nanodisc’, zegt Dirk Slotboom, hoogleraar membraaneiwit structuurbiologie aan de Rijksuniversiteit Groningen. ‘En dan ben je een deel van die instabiliteit van membraaneiwitten kwijt.’ Het lukt structuurbiologen dan ook steeds beter om membraaneiwitten stabiel voor de camera te krijgen.
‘Er ontbreekt nog heel veel kennis’
Anastassia Vorobieva, VIB-VUB
De echte stroomversnelling kwam met de ontwikkeling van machinelearning. Met AlphaFold als een van de bekendste, maar lang niet enige tool. Beeldverwerking vormt immers een groot onderdeel van structuurbepalingen, en laat machinelearning daar nou net heel goed in zijn. ‘Dat maakt dat de structuren nu makkelijker zijn op te lossen’, zegt Rouslan Efremov, groepsleider structural biology of molecular machines bij het VIB-VUB Centrer for Structural Biology.
Dat begint al bij het vastleggen van de elektronendichtheden van het eiwit met Cryo-EM onder verschillende hoeken. Hiervoor plaatsen onderzoekers membraaneiwitten op een rooster dat gekoeld in vloeibare stikstof in de microscoop gaat. Waar voorheen onderzoekers handmatig het rooster bestudeerden om te beslissen welke locaties goede plaatjes opleveren, doet een machinelearningprogramma zoals SmartScope dit nu in een oogwenk.
‘In nanodiscs ben je een deel van die instabiliteit van membraaneiwitten kwijt’
Dirk Slotboom, RUG
Cilinder
Maar waar machinelearning echt helpt is het ontwaren van een eiwitstructuur uit de duizenden Cryo-EM opnamen. Vergelijk het met een opname van een cilinder. Het zijaanzicht is een rechthoek, maar als je van bovenaf kijkt, zie je cirkel. ‘Daarom moet je echt een hoop verschillende oriëntaties zien om de 3D vorm te kunnen begrijpen’, zegt Vorobieva.
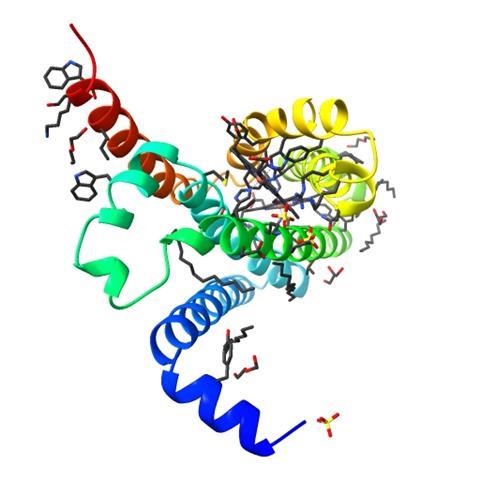
Om die puzzel op te lossen zijn grofweg drie stappen nodig. Als eerste groeperen onderzoekers de beelden met dezelfde oriëntaties. Efremov: ‘Veel van de beschikbare software lukt dit niet goed, maar op machinelearning gebaseerde programma’s zoals crYOLO en Topaz doen dit veel beter.’ En bij het wegfilteren van achtergrondruis kunnen onderzoekers een andere tool, DeepEMhancer inzetten. ‘Maar het probleem met de werkwijze van tools zoals DeepEMhancer te werk gaan is dat ze moeilijk te valideren zijn’, vervolgt Efremov.
‘Er zijn programma’s die zonder de structuur te kennen een eiwitmodel kunnen bouwen’
Rouslan Efremov, VIB-VUB
De laatste stap is het inpassen van een eiwitmodel in de experimentele data. Hier komt AlphaFold om de hoek kijken. Eiwitmodellen van programma’s zoals AlphaFold kunnen onderzoekers vergelijken met de met Cryo-EM verkregen vormen en aan de hand daarvan het voorspelde model aanpassen aan de experimentele data. ‘Maar er zijn ook programma’s die machinelearning gebruiken om aan de hand van de elektronendichthedenkaart een eiwitmodel te bouwen, zonder de structuur te kennen’, legt Efremov uit. ModelAngelo is daar een voorbeeld van.
Versnelling
Dit alles helpt structuurbiologen niet alleen om hun werk sneller te doen. Er zijn meerdere toepassingen, bijvoorbeeld het bestuderen van eiwitdynamiek. Neem transporteiwitten; die ondergaan vrij grote veranderingen in hun structuur om een molecuul van de ene naar de andere kant van het membraan te verplaatsen. Die hebben natuurlijk niet allemaal precies dezelfde houding tijdens hun Cryo-EM-opnamen.
Bij de standaardverwerking van Cryo-EM-opnamen van een eiwit nemen onderzoekers voor elke hoek waarin het eiwit is vastgelegd het gemiddelde. Dit levert een of meerdere statische vormen van het eiwit op. ‘Dus wij hebben dan snapshots van structuren’, zegt Poolman. ‘Vaak zijn die niet compleet, maar aangevuld met moleculaire dynamica-simulaties en andere tools, kunnen we een vollediger beeld krijgen, zeg maar een filmpje maken van hoe het eiwit mogelijk werkt.’
Met behulp van de machinelearningtool CryoDRGN is het nu ook mogelijk de dynamiek voor het oplossen van de structuur te zien en dat alleen aan de hand van de Cryo-EM-opnamen. Vorobieva: ‘Het gebruik is nog steeds een beetje een uitdaging, omdat je alleen de elektronendichtheden ziet bewegen.’
‘Met MD-simulaties kunnen we een filmpje maken van hoe het eiwit mogelijk werkt’
Bert Poolman, RUG
Daarnaast kan machinelearning structuurbiologen helpen bij de isolatie van membraaneiwitten wanneer er zich problemen voordoen met de stabiliteit van het eiwit. Machinelearning, legt Vorobieva uit, kan een grove voorspelling van de structuur van membraaneiwitten geven om te kunnen zien waar de problemen zitten die de stabiliteit van een eiwit verlagen. ‘Door vervolgens die problemen op te lossen lukt het om membraaneiwitten in grotere hoeveelheden te produceren en daarvan de structuur te bepalen.’
‘Wat er nu gaande is, is dat al de stappen die traditioneel handmatig en tijdrovend zijn in essentie door machinelearning worden overgenomen’, zegt Efremov. Dit heeft de bestaande aanpak enorm versneld. ‘Wat in het verleden als een uitdagend project te boek stond, is nu een gewoon standaardproject.’ Daarmee komt ruimte voor nieuwe, meer uitdagende projecten. ‘Bijvoorbeeld het precies te weten komen van hoe die structuren passen in een echte, natuurlijke of fysiologische omgeving’, besluit Slotboom.



















1 Opmerking van een lezer