Een nieuw platform met self-encoded libraries heeft het zoeken naar kandidaat-geneesmiddelen aanzienlijk vereenvoudigd. Deze methode voor het ontdekken van hits vereist geen grote biomoleculaire tags, zo tonen onderzoekers uit Leiden aan in Nature Communications. ‘Wij denken dat dit early drug discovery kan democratiseren.’
De belangrijkste uitdaging bij drug discovery is het zoeken naar nieuwe moleculen die je kunt inzetten bij farmacologische toepassingen. Naast klassieke, maar resource-intensieve industriële high-throughputscreenings bieden affiniteitsselectieplatforms een veelbelovend alternatief, waarbij DNA-encoded libraries (DEL’s) eruit springen.
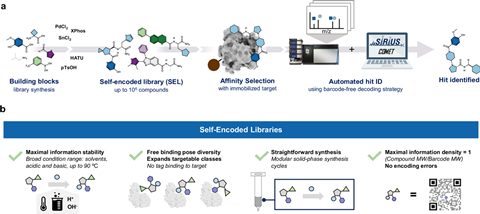
Deze affiniteitsplatforms gebruiken biologische moleculen zoals DNA of peptiden om de kleine moleculen die worden getest te labelen, waardoor je de actieve moleculen er gemakkelijk uit kunt filteren. Deze barcodes of tags kunnen echter de functie van het molecuul verstoren. Nu heeft een team onder leiding van de Universiteit Leiden en het Oncode Instituut een labelvrij platform voor het ontdekken van hits ontwikkeld, de zogenoemde self-encoded library (SEL).
Femtomol
‘Tijdens mijn eerste postdoc in medicinale chemie en chemische biologie woonde ik een conferentie bij waar ik voor het eerst kennismaakte met de technologie voor het identificeren van specifieke moleculen in grote bibliotheken’, zegt Sebastian Pomplun, universitair hoofddocent aan de Universiteit Leiden. Hij koppelde dit idee aan de manier waarop de natuur ziekteverwekkers bestrijdt, namelijk door miljoenen antilichamen te produceren, waarvan sommige zich aan een ziekteverwekker binden.
Pomplun vroeg zich af of het ook mogelijk zou zijn om met enkel synthetische stoffen te screenen. ‘Meestal is er een doelwit en een grote set verschillende moleculen voor screening. We controleren of er iets blijft plakken, spoelen alles weg en identificeren wat er overblijft.’ Het team had te maken met extreem kleine screeninghoeveelheden, soms slechts enkele femtomol per molecuul.
‘Dat lukt met massaspectrometrie als alle verbindingen verschillend zijn, waardoor de klassieke aanpak beperkt blijft tot kleine bibliotheken’, zegt Pomplun. ‘Maar met tandem-MS kunnen we de moleculen in stukjes fragmenteren, waardoor we ook veel moleculen kunnen onderscheiden die precies dezelfde massa hebben, maar verschillende structuren.’ Op deze manier kunnen ze ‘naakte’ moleculen screenen in grote bibliotheken met tot wel een half miljoen verbindingen zonder barcodes, omdat alle benodigde informatie in het molecuul zelf zit.
Shampoo
De universitair hoofddocent was erg optimistisch. ‘Ik dacht dat we dit prima zelf konden doen, dus hebben we een promovendus – Edith – aangenomen en zijn we met het project begonnen.’ Het bleek echter ingewikkelder dan verwacht, zegt Edith van der Nol. ‘We realiseerden ons dat het heel complex was. Wat echt hielp, was het aangaan van samenwerkingsverbanden met experts op het gebied van computationele massaspectrometrie-structuuraannotatie.’
Dit was een cruciale stap, omdat MS rommelig kan zijn. ‘Het pikt alles op. We kunnen zelfs verschillen zien als iemand de avond ervoor een andere shampoo heeft gebruikt’, zegt Pomplun. ‘Een van de collaborateurs, Sebastian Boecker, zag dit project als een belangrijke kans. Hij begreep de uitdaging en vond financiering om het project van de grond te krijgen.’
Decoderen
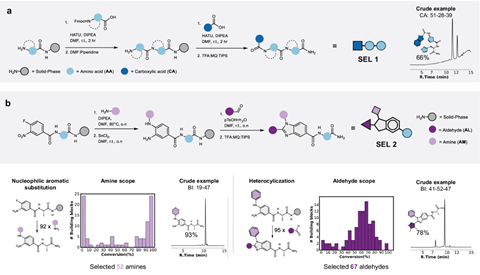
Hoe werkt SEL? Het team gebruikte solid beads om hun kleine moleculen op te bouwen, waarbij ze een split-and-pool-synthesemethode toepasten die de synthese van tienduizenden tot honderdduizenden verschillende verbindingen mogelijk maakte die uit meerdere bouwstenen bestaan. Vervolgens laten ze de moleculen reageren met een doelwit, waarbij sommige verbindingen zich daaraan binden. Tandem MS breekt de verbindingen af in delen die verschillende m/z-waarden opleveren. Met behulp van een softwareprogramma konden ze vervolgens decoderen welke verbinding zich aan het doelwit had gebonden.
Maar praatjes vullen geen gaatjes, dus gingen Pomplun, Van der Nol en hun collega’s aan de slag met een praktisch voorbeeld. ‘We moesten uitgebreide benchmarking doen’, zegt de promovendus. ‘We hadden een modelproteïne nodig om aan te tonen dat ons platform correct functioneerde en dat we de binders konden identificeren.’ In samenwerking met het Leids Universitair Medisch Centrum richtten ze zich op flap-endonuclease 1 (FEN1). Dit DNA-verwerkende enzym zou je niet kunnen bestuderen met DEL’s. Pomplun: ‘Voor het eerst zijn we in staat geweest om dit soort doelwitten te screenen.’
Uitdagend
Hoewel de methode uiteindelijk succesvol zou blijken, waren zowel Pomplun als Van der Nol verrast door hoe ingewikkeld het in het begin was. ‘Het was een hele uitdaging om een zuivere synthese te bereiken, te decoderen en wederzijds begrip te kweken met onze informaticacollega’s’, zegt Pomplun. ‘Maar toen alles op zijn plek viel in een test met onze grootste bibliotheek, zagen de gegevens er geweldig uit en bleek dat het werkte! Dit motiveerde ons om de details te verfijnen.’
SEL lijkt een goed alternatief voor de gevestigde protocollen, maar hoe vindt het zijn weg naar de laboratoria van degenen die momenteel DEL of andere methoden gebruiken? Pomplun: ‘Dat is precies wat we ons nu afvragen. Onze belangrijkste focus zal liggen op het verbeteren van ons begrip, het optimaliseren van de workflow en het verspreiden van het nieuws.’ Tijdens een conferentie in Lunteren presenteerde Van der Nol hun onderzoek en won hij de presentatieprijs. ‘Er was ook iemand van Roche aanwezig die het platform wilde bespreken’, zegt Pomplun. ‘Ik denk dat we meer interesse hebben gekregen vanuit de industrie dan vanuit de academische wereld.’
Het SEL-platform is veel eenvoudiger te implementeren dan standaardtechnieken zoals DEL of high-throughput screening. ‘Het is heel makkelijk’, zegt Van der Nol. ‘Elke masterstudent die aan het project meedeed, kon honderden, zo niet duizenden verbindingen produceren. Dit maakt het interessant voor de industrie, omdat we niet concurreren met bestaande methoden, maar deze aanvullen.’ Pomplun gelooft dat ‘ons platform early drug discovery zou kunnen democratiseren.’
Van der Nol, E. et al. (2025) Nat. Commun. 16, DOI: 10.1038/s41467-025-65282-1





















Nog geen opmerkingen