Paracetamol wordt, in tegenstelling tot andere geneesmiddelen,wél wordt afgebroken door bacteriën. Nijmeegs onderzoek geeft nu meer houvast over hoe ze dat doen en wat ons dat leert over bacteriële evolutie.
Jaarlijks plassen we met z’n allen een keur aan (actieve bestanddelen van) geneesmiddelen weer uit en die komen zo in het afvalwater terecht. Omdat het hier om niet-natuurlijke stoffen gaat, zijn ze vaak zeer moeilijk af te breken door de bacteriën in afvalwaterzuiveringsinstallaties. Andere verwijderingsmethodes zijn, vanwege de lage concentraties en de variatie aan verbindingen, zeer kostbaar. Als die al voorhanden zijn. Het gevolg is dat deze verontreinigingen in het milieu belanden en zich daar ophopen. Dat is een probleem voor allerlei levensvormen, inclusief de mens omdat deze stoffen uiteindelijk ook weer in het drinkwater opduiken.
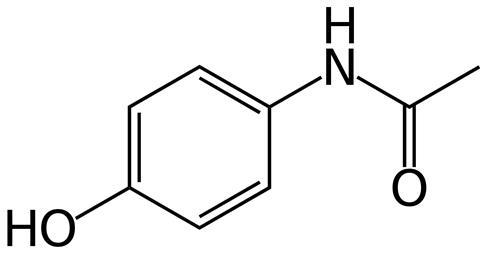
Een gunstige uitzondering is N-acetyl-p-aminofenol of acetaminofen, dat de meesten van ons kennen als paracetamol. Deze veelgebruikte pijnstiller wordt namelijk wel volledig afgebroken tijdens het waterzuiveringsproces, maar welke (bacteriële) genen en eiwitten hiervoor zorgen was nog een vraag.
Microbiologen van de Radboud Universiteit besloten daarom om slib uit een afvalwaterzuiveringsinstallatie van een ziekenhuis te laten groeien op paracetamol als enige koolstofbron.
Amidases
Door vervolgens deze verrijkte (want wie paracetamol kan afbreken, groeit sneller) bacteriële gemeenschap te analyseren met metagenomics en metatranscriptomics analyses lieten ze zien dat verschillende bacteriën betrokken zijn bij (een deel van) de paracetamol-degradatie. Kweekjes op paracetamol-agarplaten leverden twee Pseudomonas-isolaten. Een snelgroeiende stam die paracetamol omzet naar 4-aminofenol en een langzaam groeiende stam die in meer dan 90 dagen paracetamol volledig afbreekt zonder duidelijke intermediairen. Beide stammen hebben hoge expressieniveaus van een amidase, een enzym dat amides hydrolyseert.
Deze amidasegenen troffen de onderzoekers echter niet aan in het metagenoom van het slib uit de bioreactor, wat er volgens hen op duidt dat er nog meer nieuwe, niet-gekarakteriseerde amidases zijn betrokken bij het degradatieproces. Daarnaast zag het team hoge expressieniveaus van deaminase en dioxygenase genen in zowel het bioreactor metagenoom als in de twee Pseudomonas-genomen. Veel van deze genen zijn ook onderdeel van de degradatieroutes van aromatische aminozuren.
Evolutie
Bacteriën maken, zo lijkt het, daarmee handig gebruik van de bestaande capaciteit van verschillende enzymen om ook een niet-natuurlijke stof deels af te breken. Daarnaast is er ook sprake van cross-feeding; verschillende soorten bacteriën ‘eten’ elkaars afbraakproducten waardoor een complex molecuul uiteindelijk toch volledig wordt afgebroken, terwijl geen enkele bacterie dat helemaal zelf kan.
De genen die coderen voor de amidases bevonden zich op het genoom vaak in de buurt van zogeheten mobiele genetische elementen; stukken genetische informatie die makkelijk worden uitgewisseld. Dit suggereert dat horizontal gene transfer een rol speelt in de evolutie van deze bacteriën om ook paracetamol te kunnen verwerken. Volgens het Nijmeegse team bieden deze resultaten nieuwe inzichten in hoe bacteriën het vermogen verwerven om farmaceutische stoffen af te breken. Tegelijkertijd laat de grote verscheidenheid aan betrokken enzymen zien hoe complex dit proces is.















Nog geen opmerkingen