Ondanks dat lignine al best een tijd wordt geanalyseerd, is het nog een hele uitdaging om de vertakkingen goed in kaart te brengen. Een Amerikaans-Vlaams team vond een massaspectrometrie-methode waarmee dit een heel stuk makkelijker is geworden.
Lignine, een hoofdbestanddeel van de celwanden van plantenweefsels zoals hout, is een aromatisch polymeer dat deels lineair is en deels vertakt. Het is opgebouwd uit vooral guaiacyl- en syringyl-eenheden, maar kan ook andere soorten fenolische eenheden bevatten. Wil je weten hoe zo’n polymeer eruit ziet, dan kun je het in stukjes breken en analyseren met massaspectrometrie (MS). ‘Lignine met MS analyseren is al vrij lang aan de gang’, vertelt Kris Morreel, gastprofessor aan de UGent en senior scientist bij RIC Group. ‘Maar sommige oligomere fragmenten die weinig voorkomen zijn lastig te identificeren. Toch is die analyse belangrijk om vertakkingen in de sequentie aan te duiden.’ Als je niet weet wat je in handen hebt, dan kun je er natuurlijk ook niks mee.
De identiteit van de meeste ligninebrokken kun je met LC-MS wel achterhalen, maar dimeren afkomstig van vertakkingspunten zijn lastiger. ‘Dat komt omdat die dimeren lastig te verbreken bindingen heeft’, legt Morreel uit. ‘Het gaat om 5-5 en 4-O-5, respectievelijk een bifenyl met een covalente C–C-binding en een bifenylether. Beide bindingen breken niet graag.’
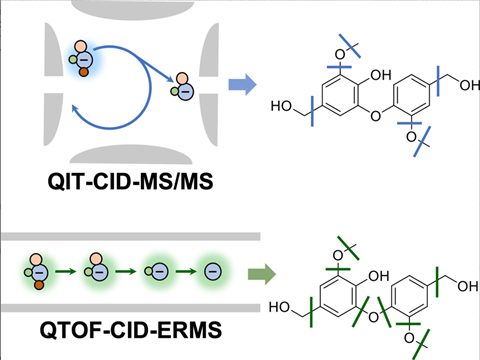
Morreel werkte samen met een team van Amerikaanse wetenschappers om de fragmenten van juist die dimeren met een speciale vorm van MS op te helderen. ‘We vergeleken data van quadrupole ion trap [QIT, red.] met quadrupole time-of-flight [QTOF, red.] massaspectrometrie die net anders werken.’ (Zie kader QTOF vs. QIT beneden.) Kort gezegd gaf de QTOF-variant meer fragmentaties en dus meer data. ‘Finaal konden we zo een onderscheid maken tussen 5-5 en 4-O-5, waar dit voorheen niet mogelijk was. We zagen namelijk dat de 4-O-5 wél fragmenteert, maar de 5-5 niet. Die kennis kunnen we dan in de toekomst incorporeren om een vertakkingspatroon te achterhalen.’
Met deze methode kun je meer en meer data genereren over lignine, ook over de vertakkingen. Morreel: ‘Dit werk kun je nu gebruiken om met algoritmes te gaan kijken naar vertakkingspunten van langere oligolignolen, langere lignineketens dus. Als we dan een databank aanleggen van geïdentificeerde oligolignolen en er vervolgens machine learning op loslaten, dan kun je toewerken naar wat richtlijnen voor de interpretatie van de data.’
Dong, X., Mayes, H., Morreel, K. et al. (2022) ChemSusChem e202201441, doi.org/10.1002/cssc.202201441
QTOF vs. QIT
QTOF kun je vergelijken met een paar aaneengeschakelde kamers waar de verschillende stappen plaatsvinden. Het parent ion dat vanuit de eerste cel binnenkomt in de collisiecel moet voldoende energie hebben om die ook weer te verlaten, zodat dochterionen in de derde cel kunnen worden geanalyseerd. Op deze manier krijg je nog relatief veel fragmentatie.
Waar je QTOF een tandem in space kunt noemen, is QIT een tandem in time. Het is een soort eivormige holte waarin ionen binnenkomen. Als dat ei zich gevuld heeft, worden de ionen eruit gepusht. Er is minder fragmentatie en dus minder data, maar het voordeel is dat je steeds één ion kunt isoleren en verder fragmenteren, waardoor je info krijgt over dochter- en kleindochterionen.
Die informatie krijg je niet zo makkelijk uit een QTOF, die moet je zelf afleiden uit de data die je verzamelt. Die verdere fragmentatie gebeurt namelijk wel, maar in één keer. Zo zijn QIT en QTOF eigenlijk complementair. Wil je veel informatie? Gebruik dan QTOF. Wil je een structuur ophelderen door toe te spitsen op het fragmentatiemechanisme, dan is de QIT soms beter omdat je echt per ion kunt kijken.
















Nog geen opmerkingen